Biodiversité Temps de lecture 6 min
FROGS : une grenouille à l’assaut des champignons
Publié le 28 septembre 2021
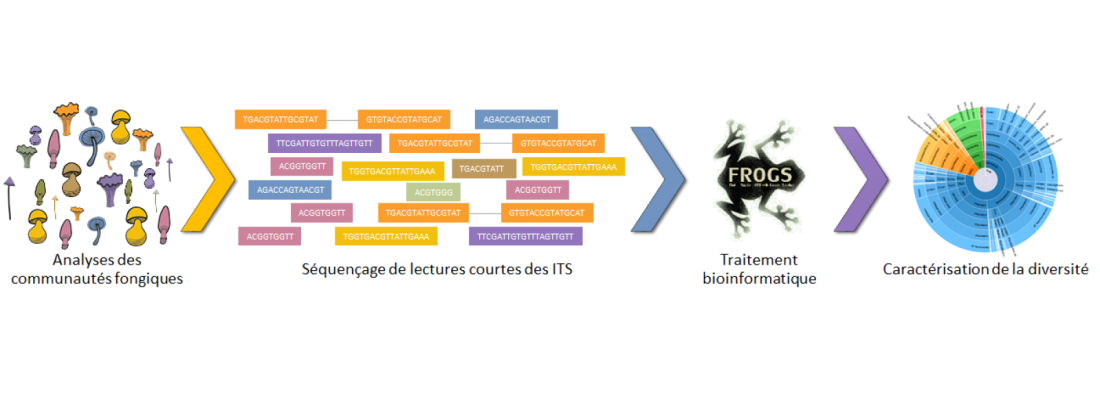
Selon les estimations actuelles, 1,5 à 5,1 millions d’espèces composent le règne des Fungi qui regroupe les levures, les moisissures et les champignons. Leur caractérisation est essentielle car toutes ces espèces ont des rôles écologiques et économiques déterminants ; elle se heurte néanmoins à des verrous méthodologiques. Le métabarcoding est une méthode largement utilisée aujourd’hui par les biologistes pour identifier facilement et à faible coût, les microbes d’un environnement. Le principe consiste en un séquençage d’une région génomique suffisamment discriminante pour identifier les différents organismes d’un milieu.
Cependant, la méthode du métabarcoding ne permettait pas, jusqu’à présent, d’utiliser correctement les marqueurs ITS (internal transcribed spacers), qui sont communément employés pour identifier les espèces fongiques. La principale difficulté avec les ITS provient du polymorphisme de leur taille. Les amplicons ITS sont des séquences d’ADN obtenues par amplification PCR. Le problème est que les séquences ainsi amplifiées varient considérablement en longueur. Or, la méthode du metabarcoding reposait jusqu’à maintenant sur des techniques de lectures de séquences courtes (deux lectures de 150 à 300 nucléotides pour un même fragment d’ADN). La plupart des outils traitent alors soit seulement les lectures qui se chevauchent, soit seulement une des deux lectures, ou font une combinaison des deux. Dans tous les cas, l’information issue du séquençage n’est jamais entièrement traitée et une fraction des espèces fongiques du milieu n’est jamais correctement identifiée.
FROGS est un outil d’analyse utilisant la technique du métabarcoding qui permet, depuis 2015, la caractérisation des microbiotes grâce au traitement des amplicons marqueurs, notamment des bactéries. Il offre désormais une solution innovante pour le traitement des lectures non-chevauchantes, par exemple les ITS, sans l’inconvénient de perte d’information mentionnée précédemment. FROGS est en outre un outil convivial, doté d’une interface graphique, qui offre le choix d’une utilisation en ligne de commande ou via une interface web (Galaxy) ; il permet ainsi à l’utilisateur d’obtenir rapidement et facilement une image précise des communautés fongiques présentes dans les milieux étudiés. En comparaison des outils les plus populaires, mais ne disposant pas de cette capacité de lecture innovante (DADA2, QIIME2, USEARCH), FROGS produit très peu de faux positifs ou de faux négatifs ; il a une excellente sensibilité et une précision élevée.
Cette nouvelle version de FROGS est le fruit d’une collaboration entre les laboratoires INRAE GABI et MaIAGE de Jouy-en-Josas et GenPhySE de Toulouse. Elle a été décrite dans un article scientifique paru dans Briefings in Bioinformatics et est notamment disponible sur les plateformes de http://bioinfo.genotoul.fr/ et https://migale.inrae.fr/. FROGS est utilisable pour tout type d'amplicons chevauchants i.e. 16S, 18S, coi, rpob… ou non chevauchants i.e. ITS1, ITS2, D1-D2, rpb2…
Les codes de FROGS sont accessibles et disponibles sur https://github.com/geraldinepascal/FROGS et via conda : https://anaconda.org/bioconda/frogs
Pour plus d’informations, rendez-vous sur le site internet de FROGS : http://frogs.toulouse.inrae.fr
Contact : frogs-support@inrae.fr
Maria Bernard, Olivier Rué, Mahendra Mariadassou, Géraldine Pascal, FROGS: a powerful tool to analyse the diversity of fungi with special management of internal transcribed spacers, Briefings in Bioinformatics, 2021; bbab318
https://doi.org/10.1093/bib/bbab318
