Les recherches du département Génétique animale


Les recherches du département couvrent 3 champs thématiques qui pavent l’ensemble des approches disciplinaires et fronts de science mis en œuvre. Elles s’organisent autour de 4 grandes priorités de recherche qui structurent le schéma stratégique 2021-2025 en réponse aux grands enjeux pour l’élevage (transition agroécologique, changement climatique, bien-être animal).
Les champs thématiques (CT) : Diversité des génomes : fonctionnement et dynamiques évolutives (CT1) ; Variabilité et élaboration des phénotypes : architecture génétique, interactions et transmission (CT2) ; Populations animales pour des élevages durables : gestion de la diversité et amélioration génétique (CT3).
Les priorités : (1) Comprendre et modéliser la variabilité génétique des caractères clés pour la transition agroécologique des élevages ; (2) Mobiliser la diversité génétique animale au service de systèmes d’élevage durables ; (3) Comprendre et modéliser la dynamique et la régulation de l’expression des génomes ; (4) Accélérer les innovations méthodologiques en génétique en mobilisant technologies du numérique et sciences des données.
TROIS CHAMPS THEMATIQUES
Pour étudier et comprendre les mécanismes génétiques et non génétiques à l’origine de la variabilité des caractères d’intérêt en élevage, les différentes échelles d’organisation du vivant (génome, animal, population) sont prises en compte. Les connaissances produites sont combinées dans des approches de biologie prédictive afin de proposer des méthodes permettant de préserver et de valoriser durablement la diversité génétique présente dans les populations animales.
CT1 - Diversité des génomes : fonctionnement et dynamiques évolutives
Les objectifs sont (i) de décrire de manière aussi complète que possible la diversité des génomes nucléaires et mitochondriaux, épigénomes (et métagénomes[1]) de nos espèces d’intérêt ; (ii) de comprendre leur fonctionnement, en attribuant une signification biologique aux éléments non codants et en décortiquant les mécanismes moléculaires qui régulent leur expression et sont à l’origine de la variabilité des phénotypes, (iii) de comprendre et interpréter leur dynamique évolutive, à la lumière des mécanismes d’évolution de leur structure et de la diversité génomique des populations animales d’aujourd’hui. La maîtrise experte des technologies de séquençage les plus récentes permet de pousser les fronts de science de ce domaine avec toujours plus d’ambition : annotation fonctionnelle des génomes, génomique régulationnelle et étude de l’impact des polymorphismes structuraux dans l’organisation 3D de la chromatine, génétique de la recombinaison, rôle des marques épigénétiques et génétique de l’épigénétique.
[1] Pour cette seule partie de caractérisation
CT2 - Variabilité et élaboration des phénotypes : architecture génétique, interactions et transmission
Les recherches portent sur l’identification des déterminants à la fois génétiques (en allant jusqu’à la mutation causale dans certains cas) et non-génétiques de la variabilité des phénotypes et sur la prédiction de leur transmission intergénérationnelle. Intégrer l’ensemble des facteurs à l’origine de cette variabilité (environnement, interactions hôte-microbiote(s), marques épigénétiques, apprentissage, …), les corrélations génétiques entre caractères et le contrôle génétique des compromis entre fonctions pour modéliser et prédire les phénotypes et leur évolution est un nouveau front de science. Avec la transition agroécologique, l’étude des interactions génotype x environnement (incluant les pratiques d’élevage) prend une importance croissante. Les travaux bénéficient (i) des avancées des « -omiques », qui fournissent des phénotypes intermédiaires souvent plus représentatifs de l’effet des polymorphismes d’intérêt et (ii) des outils numériques pour le phénotypage (capteurs, robots, …). Les fronts de science sont notamment d’ordre méthodologique : intégration dans les modèles de prédiction de données de plus en plus nombreuses, hétérogènes et de propriétés statistiques complexes. Le recours à des méthodes de phénotypage peu invasives à des méthodes alternatives à l’expérimentation animale, est un autre enjeu fort. Les efforts portent sur les caractères clés de la durabilité (santé, robustesse, efficience, comportements).
CT3 - Populations animales pour des élevages durables : gestion de la diversité et amélioration génétique
Il s’agit de proposer des outils, des méthodes et des stratégies pour préserver, valoriser et exploiter durablement la diversité génétique intra et inter populations, et ce, dans des environnements variés et changeants. Les fronts de science sont la prédiction génomique et son enrichissement avec des informations biologiques (interactions, mutations causales, réseaux de gènes, …) afin d’améliorer sa précision, sa persistance entre générations et sa transposabilité (entre populations, voire entre espèces), et l’optimisation des programmes d’amélioration en fonction du contexte biologique et socio-économique.
QUATRE PRIORITES SCIENTIFIQUES
La diversité génétique des populations animales est une ressource précieuse pour le développement de systèmes d’élevage innovants et durables. Notre ambition est de produire des connaissances et des outils génériques mobilisables pour différents scénarios d’évolution des systèmes d’élevage, y compris ceux impliquant de réelles ruptures.
Nos travaux s’inscrivent dans le contexte du développement des démarches participatives, de montée en puissance du numérique et des méthodes d’intelligence artificielle et de la science ouverte.
- Comprendre et modéliser la variabilité génétique des caractères clés pour la transition agroécologique des élevages
Les élevages de demain, plus ouverts et moins standardisés, feront davantage appel à la capacité des animaux à faire face à des perturbations sanitaires ou environnementales, tout en conservant leurs aptitudes productives. Dans ce contexte, les priorités sont de caractériser le déterminisme de nouveaux caractères pour préserver la santé et favoriser le bien-être des animaux, de renouveler les approches pour améliorer l’efficience productive et environnementale, de comprendre et modéliser les interactions entre caractères pour favoriser la résilience et l’adaptation des animaux, en intégrant l’étude du rôle des microbiotes et des marques épigénétiques dans la variabilité et la transmissibilité des caractères.
- Mobiliser la diversité génétique animale au service de systèmes d’élevage durables
Systèmes de production, conduites d’élevage et caractéristiques des animaux doivent être réfléchis simultanément, dans des démarches de compromis et d’articulation des leviers pour répondre aux exigences de la multiperformance globale. De nouvelles manières de mobiliser la diversité génétique doivent donc être explorées, que ce soit dans le choix des populations (races locales, croisements, mélanges de populations ou d’individus de caractéristiques diverses dans les troupeaux, populations synthétiques, … ) ou dans la définition d’objectifs d’amélioration génétique des populations qui permettent de combiner les objectifs de la durabilité et d’adaptation des animaux et des élevages à des environnements en évolution. Les programmes de gestion et d’amélioration génétique eux-mêmes doivent être adaptés pour répondre aux besoins de systèmes plus diversifiés.
- Comprendre et modéliser la dynamique et la régulation de l’expression des génomes
La connaissance de la structure et la compréhension du fonctionnement et de la régulation des génomes animaux sont à la fois un enjeu de connaissance et un outil indispensable pour l’exploration de l’architecture génétique des caractères complexes, et pour l’utilisation et la gestion de la diversité génétique des populations animales. Nous mettrons notamment l’action sur l’annotation fonctionnelle des génomes.
- Accélérer les innovations méthodologiques en génétique en mobilisant technologies du numérique et sciences des données
Les évolutions technologiques dans le domaine de la génomique au sens large ont été fulgurantes, qu’il s’agisse des approches qu’elles proposent, des débits qu’elles procurent ou de leur coût de plus en plus accessible. Les nouvelles générations de séquençage (NGS) donnent accès à toute la variabilité de l’ADN du génome, à la mesure de l’expression des gènes, à l’épigénome, ou encore au génome du microbiote des animaux dans des organes variés. Toutes ces informations, couplées aux données phénomiques apportées par les capteurs et autres instruments de mesure, sur l’animal et son environnement au sens large, vont permettre de progresser dans plusieurs domaines pour (i) mobiliser le numérique pour un phénotypage haut débit et sur de grandes cohortes ; (ii) développer de nouvelles approches de biologie intégrative pour exploiter les opportunités de la phénomique et (iii) enrichir les méthodes de prédiction et de sélection avec des informations biologiques.
NOS PARTENARIATS ACADEMIQUES
Les équipes du département ont développé un écosystème partenarial riche, qui leur permet de développer les approches multidisciplinaires permettant de répondre aux questions complexes posées aux élevages d’aujourd’hui.
Les collaborations avec les autres départements INRAE sont nombreuses (nous collaborons avec 10 d’entre eux). La plupart de nos unités sont des UMR avec les écoles d’agronomie (AgroParisTech, Institut Agro Rennes, INP-ENSA Toulouse et école de Purpan) ou vétérinaires (ENVT, Toulouse) et nous collaborons avec des équipes Inserm et CNRS sur quelques sujets spécifiques.
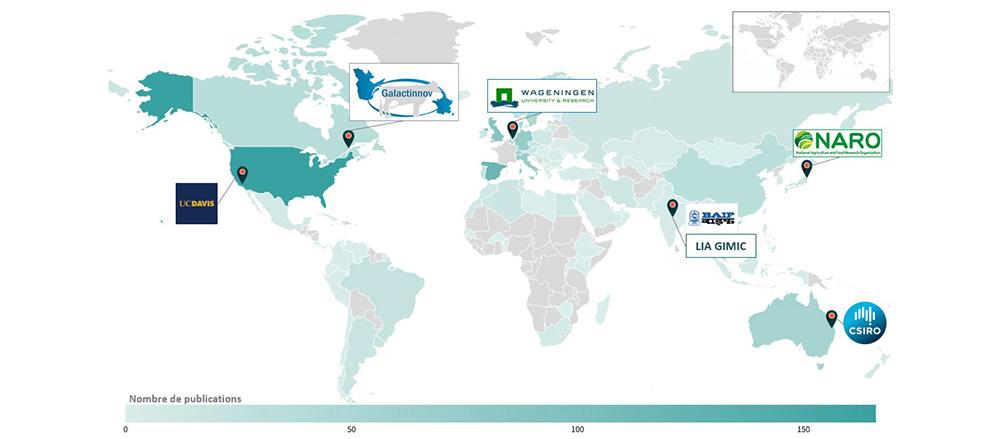
Nos collaborations en Europe et à l’international
Plus de la moitié de nos publications sont cosignées avec un partenaire étranger. Nous avons des partenariats en Europe, en Amérique du Nord et en Amérique Latine, en Inde, au Japon, en Australie, et quelques collaborations en Afrique.
Lien vers la collection des publications du département déposées dans l'archive ouverte HAL-INRAE
Quelques projets emblématiques

Lien interne
L’annotation des génomes animaux domestiques made in FranceCOMMUNIQUE DE PRESSE - Annoter le génome d’une espèce d’intérêt, c’est établir le rôle des différents éléments de celui-ci. Cette cartographie fonctionnelle permet ensuite de mieux comprendre les liens entre les gènes et les phénotypes (1). Des chercheurs d’INRAE ont lancé le projet pilote français FR-AgENCODE afin d’enrichir l’annotation du génome de quatre espèces animales d’élevage (vache, poule, chèvre et porc).

Lien interne
SMARTER H2020 : la sélection génétique des petits ruminants pour un élevage durableDans le cadre du programme européen H2020, le projet Small Ruminants breeding for Efficiency and Resilience (SMARTER) des équipes du laboratoire Génétique Physiologie et Systèmes d’Elevage (GENPhySE) du centre INRAE Occitanie-Toulouse, a été retenu et obtenu un financement de 7 millions d’euros sur 4 ans. Ce projet d’envergure étudie l’amélioration de la sélection génétique afin d’accroître la résilience et l’efficience des petits ruminants dans leur milieu d’élevage.

Lien interne
H2020 GEroNIMO : penser l’élevage de demainDans le cadre du programme européen Horizon 2020, le projet "Genome and Epigenome eNabled breedIng in MOnogastrics" (GEroNIMO), des équipes des laboratoires Génétique Physiologie et Systèmes d’Elevage (GenPhySE) du centre INRAE Occitanie-Toulouse et Génétique Animale et Biologie Intégrative (GABI) à Jouy-en-Josas, ont obtenu un financement de 7 millions d’euros sur 5 ans.
