Alimentation, santé globale Temps de lecture 2 min
Déchiffrer le potentiel des écosystèmes microbiens grâce à une nouvelle approche microfluidique
Publié le 20 octobre 2020
Les écosystèmes microbiens représentent une mine d’or pour la prospection de nouvelles fonctions d’intérêt biotechnologique. Pourtant, la majorité des espèces qui composent ces communautés complexes sont difficiles, voire impossibles à étudier.
Une nouvelle approche technologique
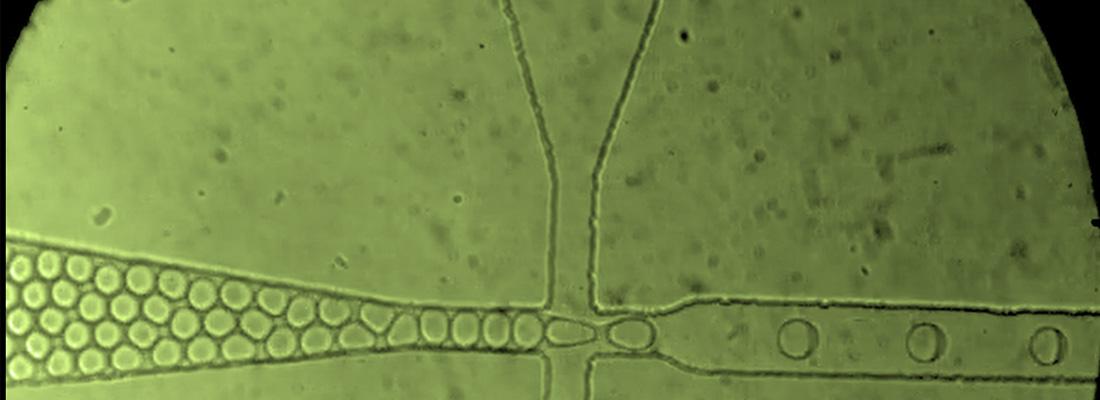
Dans le cadre des projets européens Catsys et Metafluidics, les chercheurs du Toulouse Biotechnology Institute et de l’Université de Cambridge ont développé une nouvelle approche microfluidique* pour explorer sans limites le potentiel de ces écosystèmes microbiens. Cette technique de criblage à ultra-haut-débit est miniaturisée à l’échelle du picolitre, dans des gouttelettes de 18 µm de diamètre. Au moins 1000 fois plus rapide et moins coûteuse que les technologies conventionnelles, elle permet d’explorer la diversité fonctionnelle de milliers de génomes bactériens en seulement une heure de travail.
*La microfluidique est la science et la technologie des systèmes manipulant des fluides et dont au moins l'une des dimensions caractéristiques est de l'ordre du micromètre.
Pour quelles applications ?
Ces approches miniaturisées sont compatibles avec la découverte et l’ingénierie d’une très grande variété d’enzymes et de voies métaboliques d’intérêt pour la santé humaine et animale, ou, par exemple, pour la décontamination des écosystèmes pollués. Cette technologie a d’ailleurs déjà permis d’identifier de nouvelles interactions entre l’homme et son microbiote, en particulier dans le contexte de maladies inflammatoires chroniques de l’intestin.
Des perspectives encourageantes
Ces développements offrent de larges perspectives pour la métagénomique fonctionnelle, c’est-à-dire l’étude des fonctions codées par l'ensemble de l'ADN des microorganismes issus d'environnements complexes (ex : intestin, océan, sol, air, etc.). Ils ouvrent ainsi une nouvelle ère pour la découverte de nouvelles enzymes et autres voies de synthèse de molécules d’intérêt biotechnologique.
Investigating host-microbiome interactions by droplet based microfluidics Alexandra S. Tauzin, Mariana Rangel Pereira, Liisa D. Van Vliet, Pierre-Yves Colin, Elisabeth Laville, Jeremy Esque, Sandrine Laguerre, Bernard Henrissat, Nicolas Terrapon, Vincent Lombard, Marion Leclerc, Joël Doré, Florian Hollfelder & Gabrielle Potocki-Veronese Microbiome volume 8, Article number: 141 (2020) https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-020-00911-z
