Bioéconomie Temps de lecture 3 min
Comprendre comment les bactéries s’adaptent à leur environnement
Publié le 12 février 2021
Quand une bactérie contrôle la dégradation de ses ARNm pour s’adapter
Une équipe de chercheurs du centre INRAE Occitanie-Toulouse, en collaboration avec l’Inria de Grenoble a exploré les mécanismes de régulation de la dégradation des ARNm, en se basant sur l’exemple de l’adaptation de la bactérie Escherichia coli à son environnement. « Que ce soit dans les milieux naturels comme synthétiques, E. coli doit faire face à des changements, tels que la disponibilité des sources de carbone », explique Muriel Cocaign-Bousquet, directrice de recherche INRAE. Pour s’adapter, la bactérie met en œuvre une régulation de la dégradation des ARNm comme un moyen de favoriser ou, au contraire, de défavoriser l’expression de certains gènes afin d’optimiser son fonctionnement».
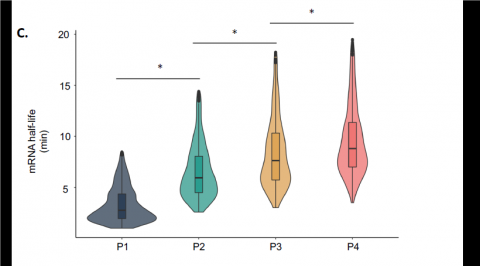
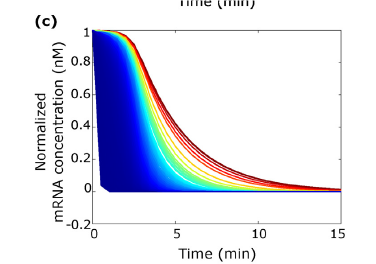
Comprendre l’adaptation des bactéries pour obtenir des souches innovantes
Grâce à des méthodes de biologie à haut débit et des approches de modélisation, les chercheurs du Toulouse Biotechnology Institute peuvent mesurer individuellement la dégradation de plus de 4000 ARNm de la cellule d’E. coli. Cela leur a permis de comprendre comment E. coli régule de façon différente la dégradation de certains ARNm en fonction de la nature et de la concentration de la source de carbone et d’identifier 18 ARNm majeurs pour l’adaptation de E. coli à son environnement. Les chercheurs ont également caractérisé les enzymes impliquées dans la dégradation des ARNm et découvert un nouveau mécanisme de régulation lié à une compétition entre des milliers de molécules d’ARNm pour se lier à une enzyme, l’endoribonucléase RNase E.
Cette meilleure compréhension des processus d’adaptation d’E. coli ouvre des perspectives d’ingénierie du cycle de vie des ARNm pour améliorer les performances de cette bactérie en biotechnologie. En jouant sur les régulations des mécanismes et des cinétiques de dégradation des ARNm, il serait en effet possible de mieux contrôler son expression génétique. « Ces travaux nous permettent d’envisager des souches innovantes d’E. coli, mieux adaptées à l’utilisation de nouvelles sources de carbone pour la synthèse de protéines et de métabolites d’intérêt pour l’industrie », conclut Muriel Cocaign-Bousquet.
Morin M, Enjalbert B, Ropers D, Girbal L, Cocaign-Bousquet M. Genomewide Stabilization of mRNA during a "Feast-to-Famine" Growth Transition in Escherichia coli. mSphere. 2020 May 20;5(3):e00276-20. doi: 10.1128/mSphere.00276-20.
Etienne TA, Cocaign-Bousquet M, Ropers D. Competitive effects in bacterial mRNA decay. J Theor Biol. 2020 Nov 7;504:110333. https://doi.org/10.1016/j.jtbi.2020.110333 Epub 2020 Jun 29. PMID: 32615126.
