Agroécologie Temps de lecture 7 min
ABC des techniques de modifications ciblées du génome
Publié le 26 avril 2019

Jusqu’aux années 2010, l’insertion d’un transgène se faisait au hasard dans le génome, avec la possibilité de différents cas de figure non désirés : remaniements du transgène, réarrangements du génome autour du site d'insertion, insertions dans des gènes actifs, insertion dans de la chromatine « silencieuse » défavorable à l’expression du transgène, etc. Ce caractère aléatoire de l’insertion rendait nécessaire l’observation d’un grand nombre de plantes transformées et la caractérisation a posteriori de l’insertion par séquençage pour obtenir une plante transformée satisfaisante.
Depuis quelques années, il existe des techniques qui permettent de cibler précisément le site du génome sur lequel on veut intervenir, que ce soit pour insérer un transgène ou bien pour modifier un gène sur place (gene editing).
Plusieurs générations de techniques sont apparues successivement. Toutes reposent sur des nucléases capables de reconnaitre spécifiquement une séquence d’ADN donnée et de provoquer une coupure unique et ciblée dans le génome de la plante à modifier. Pour ce faire, on introduit dans la plante cible le gène de ces nucléases, de manière transitoire ou stable (c’est-à-dire intégré au génome). Dans ce dernier cas, ce gène est ensuite éliminé lors des croisements ultérieurs.
Une nucléase ciblée est une enzyme composée d'un domaine de liaison à l'ADN, qui détermine sa spécificité, et d'un domaine actif, qui effectue la coupure de l'ADN. Les nucléases ciblées utilisées en biotechnologies sont toutes des variantes de nucléases ou de domaines de liaison à l'ADN qui existent dans la nature. Tout le savoir-faire consiste à modifier la spécificité naturelle de ces nucléases et à faire en sorte qu'elles reconnaissent le site d'intérêt dans le génome. Cette modification de la spécificité est devenue de plus en plus facile, prédictible et bon marché en passant des méganucléases par les nucléases à doigt de zinc aux TALENs et enfin aux CRISPR-Cas9.
Les méganucléases
Les méganucléases reconnaissent comme site de coupure des séquences de très grande taille (de 12 à 40 paires de bases) dans le génome. Statistiquement une séquence de 18 paires de bases est trouvée une seule fois dans un génome de 274 millions de paires de bases, ce qui est largement supérieur à la taille du génome des plantes cultivées. Comme la centaine de méganucléases découvertes dans différents organismes ne donne qu'un choix très limité de sites cibles, les chercheurs ont modifié les gènes codant les méganucléases ou combiné des domaines de différentes méganucléases pour modifier la séquence cible reconnue. Au début purement empirique, cette ingénierie des méganucléases s'est rationnalisée à un certain degré avec l'expérience acquise. Actuellement les bibliothèques virtuelles d’enzymes disponibles sont a priori suffisamment riches pour pouvoir cibler potentiellement n’importe quel gène dans n’importe quel génome. La synthèse des méganucléases reste cependant assez lourde (environ six mois, de l’ordre de 25 000 euros) et nécessite le savoir-faire de compagnies spécialisées telles que Cellectis en France.
Les nucléases à doigt de zinc
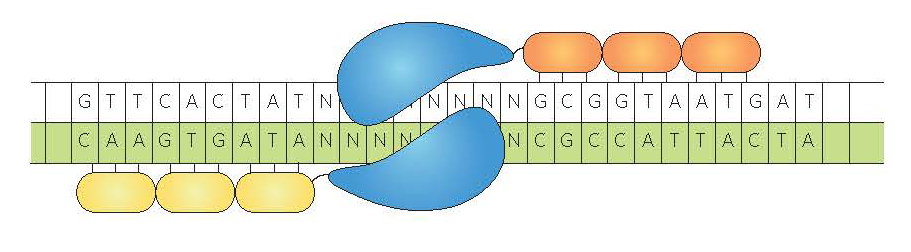
Les nucléases à doigt de zinc combinent un domaine de coupure non spécifique avec un domaine dit "doigt de zinc", qui est constitué de peptides reconnaissant chacun une séquence de trois paires de bases. Ces peptides sont choisis en fonction de la séquence cible et assemblés l'un derrière l'autre en domaine. Comme la spécificité de ces peptides n'est pas très bonne et influencée par l'assemblage, il faut en général tester plusieurs dizaines de nucléases, qui théoriquement reconnaissent toutes la séquence cible d'intérêt, pour en trouver une qui a une spécificité acceptable. Par ailleurs, pour obtenir une coupure complète de l'ADN, il faut toujours combiner deux nucléases à doigt de zinc, car chacune ne coupe qu'un seul brin de l'ADN.
Les TALENs
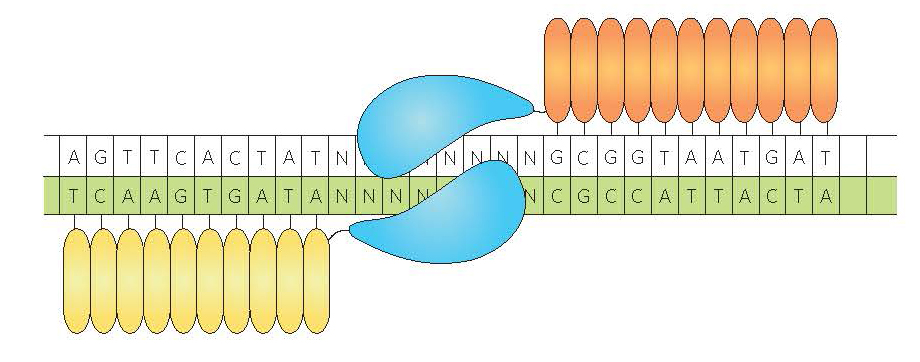
Décrites pour la première fois en 2009, les TALENs (Transcription activator-like-effector-nucléases) sont également des nucléases artificielles composées d’un domaine de liaison à l’ADN (TALE) et d’une nucléase capable de couper l’ADN. Le domaine de liaison à l’ADN est fabriqué sur la base des connaissances acquises sur un facteur de transcription bactérien, formé de plusieurs peptides de 34 acides aminés assez conservés, à l’exception des deux acides aminés en position 12 et 13 qui reconnaissent spécifiquement un ou deux des quatre nucléotides de l’ADN. Dès lors, si la séquence d’ADN que l’on veut couper comporte 20 paires de bases, on synthétisera la TALEN adéquate en assemblant, comme dans un jeu de lego, les 20 peptides de 34 acides aminés qui reconnaissent successivement chacune des 20 paires de bases. Comme dans le cas des nucléases à doigt de zinc, il faut deux TALEN pour une coupure des deux brins de l'ADN. Comme la fiabilité du code de reconnaissance entre peptides et ADN est très bonne, en pratique, plus d'un couple TALEN sur deux coupe la séquence cible avec une bonne efficacité. Par la fiabilité accrue du design, cette technique a été la première accessible à des laboratoires non-spécialistes, même si le clonage et le séquençage des répétitions demande une certaine technicité.
Le système CRISPR-Cas9
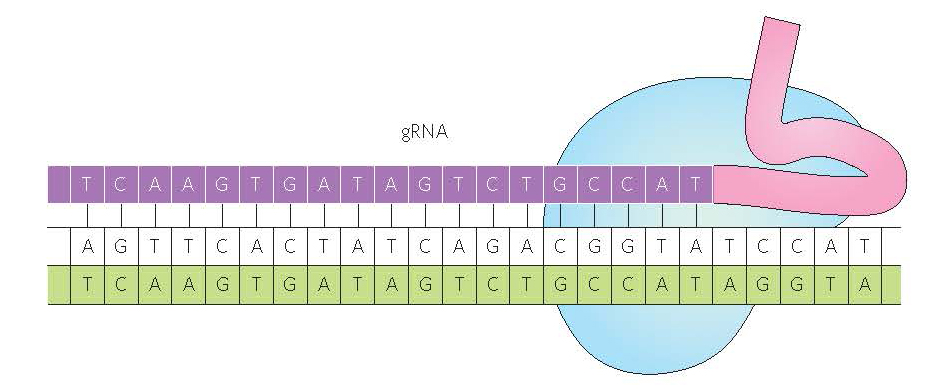
Le système CRISPR-Cas9 (Clustered-regularly-interspaced-short-palindromic-repeats/Crispr associated protéines) consiste en une nucléase dont la spécificité est déterminée par un petit ARN d’une vingtaine de nucléotides, intégré dans l’enzyme. On peut dès lors fabriquer la nucléase voulue en lui associant un petit ARN homologue de la séquence d’ADN à cliver. Ce système est issu d’un mécanisme de défense immunitaire existant chez certaines bactéries pour détruire du matériel génétique étranger : dans ce cas, le petit ARN est synthétisé à partir d’un fragment de l’ADN étranger (par exemple un virus) et permet à la nucléase de cibler et de détruire cet ADN étranger.
Après la coupure ciblée de l"ADN, trois actions sont possibles : voir encadré.
Base editing (dCas9-deaminase)
Plus récemment (Komor et al., 2016. Nature 533), une autre stratégie connue sous le nom de modification de base (base editing) a été mise au point. Elle consiste à utiliser une protéine Cas9 modifiée (dCas), capable de reconnaître un site précis dans l'ADN mais incapable de le couper. Cette protéine dCas est fusionnée avec une cytidine déaminase qui transforme une base C en T (ou G en A sur le brin opposé) dans la petite fenêtre d'édition spécifiée par l'ARN guide (gRNA). On peut ainsi corriger une mutation ou muter une base sans introduire de cassure dans l’ADN.
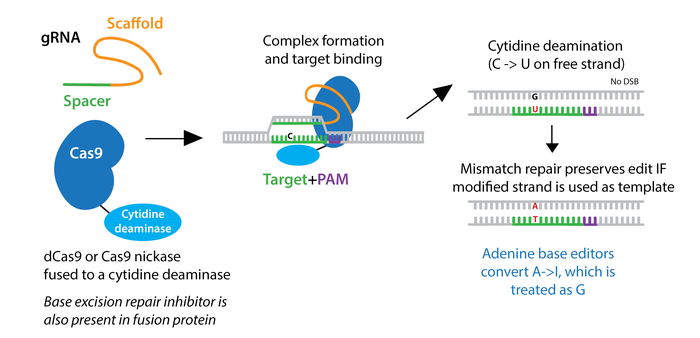
Après la coupure ciblée de l"ADN, trois actions sont possibles :
- Inactiver un gène : le KO (knock out) : on provoque une coupure dans le gène visé, ce qui déclenche la machinerie de réparation cellulaire de l’ADN. Cette réparation fonctionne dans 99 % des cas, mais dans 1 à 2 % des cas, il y a une réparation défectueuse, avec suppression ou insertion de quelques paires de base, entraînant l’inactivation du gène. L’effet obtenu est comparable à une mutation.
- Modifier un gène (gene editing, ou knock in) : on présente à l’endroit de la coupure une "matrice de réparation", une version modifiée du gène coupé, qui a été produite en laboratoire et qui est recopiée lors de la réparation grâce à un autre mécanisme de réparation, la recombinaison homologue. Cela revient à remplacer un allèle par un autre.
- Insérer un gène étranger de manière ciblée (transgénèse): la démarche est la même que pour le knock in, sauf que la matrice de réparation est un transgène flanqué de part et d'autre de séquences homologues au site de coupure. Le site de coupure ou « landing pad » (terrain d’atterrissage pour la trangénèse) peut être soit une séquence naturellement présente dans le génome, soit une séquence préalablement insérée dans le génome par transgenèse classique. L'intérêt d'une insertion ciblée est la connaissance du "landing pad", choisi ou conçu pour une bonne expression du transgène sans effet néfaste sur la plante. Dans une variante de cette technique le transgène inséré au "landing pad" original contient lui-même un deuxième "landing pad", ce qui permet l'empilement successif de deux ou plusieurs transgène au même endroit du génome ("trait stacking").

Les biotechnologies vertes reposent sur un vaste ensemble de techniques de recherche en biologie végétale, techniques de culture in vitro ou techniques moléculaires telles que mutation, transgénèse, etc., dont ce dossier donne un panorama.
