Temps de lecture 5 min
Zoom sur… les génomes et la génomique
Publié le 24 avril 2023
Le génome, un délicat équilibre entre stabilité et plasticité
Les génomes sont le support de l’information génétique de toutes les espèces et individus sur notre planète. En cela ils nous unissent tous et en même temps font que nous sommes tous différents. De l’expression de ces génomes résulte la formidable diversité d’espèces et de formes de vie, fruit de plusieurs milliards d’années d’évolution et dont nous ne connaissons finalement qu’une infime portion.
Chez nous les humains, comme chez beaucoup d’espèces, les génomes sont organisés en chromosomes et constituent un patrimoine que nous héritons de nos parents puis transmettons à nos descendants avec plus ou moins de réarrangements, de modifications ou mutations. Au cours de l’évolution des espèces, un délicat équilibre s’est établi entre stabilité du génome assurant la transmission de ce patrimoine le plus fidèlement possible et plasticité de celui-ci permettant aux espèces d’évoluer et de s’adapter.
Parfois, l’information génétique peut également se transmettre d’un individu à l’autre voire d’une espèce à l’autre sans relation de parenté directe entre elles. Ces événements dits de « transferts horizontaux » constituent un étonnant mécanisme de plasticité génomique pouvant même se produire entre espèces séparées par des centaines de millions d’années d’évolution. Plus fréquents chez les bactéries, ces transferts de gènes ont conduit par exemple aux multirésistances aux antibiotiques qui posent de nombreux problèmes en santé animale et humaine. Bien que moins courants, ces événements ont aussi eu lieu dans des génomes d’eucaryotes y compris chez les animaux où ils ont contribué à l’évolution du placenta, de l’œil ou de la capacité à parasiter les plantes.
Le séquençage de génomes, une histoire qui débute en 1976 et qui s’accélère
Le premier génome séquencé, celui du bactériophage MS2, entité virale à ARN a été publié dès 1976. Il aura fallu attendre ensuite presque 20 ans, en 1995 pour que le premier génome d’un organisme cellulaire, celui de la bactérie Haemophilus influenza soit publié. Les choses se sont ensuite accélérées grâce à la démocratisation des technologies de séquençage avec le premier génome d’un eucaryote, celui de la levure Saccharomyces cerevisiae en 1996, suivi en 1998 du génome du nématode Caenorhabditis elegans, le premier pour un animal, puis celui d’Arabidopsis thaliana, le premier pour une plante en 2000.

Le génome humain, quant à lui, a été annoncé comme séquencé une première fois dès 2001, à l’issue d’une grande bataille entre un consortium public international et l’entreprise américaine Celera Genomics, l’un faisant au final la couverture de Nature quand l’autre faisait la couverture de Science. Pour l’anecdote, alors étudiant de Master, mon laboratoire d’accueil m’avait offert ces deux numéros spéciaux de Nature et Science que je garde toujours aujourd’hui précieusement dans un tiroir de mon bureau comme une sorte de graal.
L’INRA puis INRAE ne sont pas en reste dans la grande histoire du décodage et de l’analyse des génomes. En effet, notre institut a été impliqué dans le séquençage du premier génome d’un organisme pathogène de plante, celui de la bactérie Xylella fastidiosa, sorti en 2000. Par la suite, l’INRA a également été un acteur de l’étude des génomes du premier animal d’intérêt agronomique, celui du poulet (Gallus gallus) en 2004, du premier arbre (Populus trichocarpa) ou du premier insecte pollinisateur avec l’abeille Apis mellifera, tous deux en 2006.
L’étude des génomes d’organismes liés à la santé des plantes en fil conducteur
Dans cette histoire des génomes, mon chemin a croisé celui de l’INRA une première fois entre 2006 et 2007, lorsqu’alors postdoctorant au CNRS dans le laboratoire de glyco-génomique, j’ai annoté le répertoire d’enzymes actives sur les sucres dans le génome de Laccaria bicolor, le premier pour un champignon vivant en symbiose avec les arbres. Au cours de ce post-doctorat je m’intéressais aux relations entre variations dans ces répertoires d’enzymes et mode de vie saprobe, symbiotique ou phytopathogène, via l’analyse comparative de génomes de champignons. Fin 2007, j’ai été recruté comme chargé de recherche à l’INRA pour participer à l’analyse du génome du nématode à galles Meloidogyne incognita, le premier pour un animal parasite de plantes. Nos chemins se sont alors rejoints durablement et les articles décrivant les génomes de Laccaria bicolor et Meloidogyne incognita ont tous deux étés publiés en 2008, l’un dans Nature et l’autre dans Nature Biotechnology, faisant désormais de mon histoire et de celle de l’INRA avec les génomes, une histoire commune.
Durant toutes ces années, je me suis intéressé à la comparaison de génomes, à travers le prisme de leur évolution, car « Rien n'a de sens en biologie, si ce n'est à la lumière de l'évolution », comme l’a si bien postulé Theodosius Dobzhansky, biologiste Ukrainien et théoricien de l’évolution. Identifier des singularités dans les génomes de parasites et pathogènes a été l’un de mes principaux objectifs, persuadé que ces spécificités pouvaient avoir un lien avec l’évolution de leur capacité à manipuler et exploiter un hôte. C’est ainsi que nous avons pu mettre en évidence l’importance des événements d’acquisition de gènes par transferts horizontaux dans l’évolution de la capacité des nématodes et des insectes à parasiter les plantes. Parmi les autres singularités, nous avions également remarqué qu’une part importante des gènes codés par les génomes de nématodes à galles n’avaient aucune relation d’homologie identifiable dans le reste du vivant. A l’époque cela aurait pu s’expliquer par l’absence d’espèces proches dans les bases de données de séquences. Or, nous constatons que ces gènes demeurent « orphelins » aujourd’hui malgré la présence de plus d’une centaine de génomes de nématodes y compris d’espèces proches. Ces gènes prédits dans les génomes sont en plus transcrits et on sait maintenant par exemple que la majorité des protéines effectrices du parasitisme chez les nématodes sont codées par de tels gènes dits orphelins. Leur origine évolutive reste peu voire pas connue et est devenue l’un de nos sujets d’études.
Des techniques de séquençage plus performantes et moins couteuse ouvrant de nouvelles perspectives
Les progrès récents dans les techniques de séquençage, et notamment le séquençage longues lectures (élue méthode de l’année en 2022 par Nature Methods), a propulsé la génomique vers une nouvelle ère. En effet, l’accès à ces technologies et à des coûts devenus abordables a permis d’assembler plus facilement des génomes à un niveau de résolution chromosomique. Par exemple, alors que le génome humain avait été annoncé comme séquencé dès 2001, ce n’est qu’environ 20 ans plus tard qu’un génome complétement assemblé a enfin été publié (en 2022 dans Science). Au-delà de la satisfaction d’avoir des génomes quasi ou complétement assemblés en chromosomes, que cela apporte-t-il à nos recherches ? En réalité ces nouveaux génomes de référence ouvrent de nombreuses perspectives. Ils nous permettent dorénavant, d’étudier comment l’information génétique est structurée et organisée le long des chromosomes dans les espèces d’intérêt pour INRAE.
Nous vivons en ce moment une époque absolument formidable et passionnante dans le domaine de la génomique
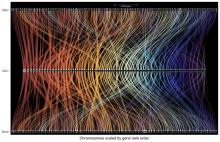
D’autre part, la comparaison de données longues lectures de différents isolats et/ou populations avec ces génomes de référence mieux assemblés nous permettent enfin d’étudier la plasticité de ces génomes au sein d’une même espèce dans toutes ses dimensions. En effet, en plus des mutations ponctuelles classiquement étudiées (SNP, SNV), les variations structurales de plus grande envergure telles que les inversions, translocations, insertions ou délétions peuvent maintenant être détectées et génotypées de manière fiable. Or, si on ne considère que les mutations ponctuelles, lorsque l’on compare deux génomes humains, nous n’avons en moyenne que 0,1% des nucléotides qui varient. En revanche, si nous considérons cette fois les variants de structure, c’est 1,5% du génome qui varie d’un individu à l’autre et donc 15 fois plus de nucléotides impactés. Ces variants de structure semblent tout aussi important que les mutations ponctuelles et leur implication dans des cancers mais aussi dans l’adaptation des espèces à leur environnement a déjà été démontrée.
Les techniques de capture de la conformation de la chromatine couplées au séquençage haut débit nous permettent quant à elles d’étudier comment l’information génétique est structurée dans l’espace au sein des cellules. Enfin, certaines techniques de séquençage longues lectures permettent de directement détecter les modifications épigénétiques des nucléotides, tandis que d’autres techniques nous informent sur les modifications des histones, impliquées dans la condensation et décondensation de l’ADN. L’ensemble de ces mutations ponctuelles ou structurales, ainsi que l’organisation du génome dans l’espace ou ses modifications épigénétiques peuvent influer sur son programme d’expression dans le temps et dans l’espace. Ce même programme d’expression (ou transcriptome) se traduisant et se manifestant quant à lui dans une diversité de phénotypes eux même soumis à la sélection naturelle au cours de l’évolution.
La génomique et l’intelligence artificielle, une alliance indispensable pour relever les nouveaux défis
Ces différentes avancées technologiques nous permettent d’appréhender les relations entre génomes et phénomes (l’ensemble des phénotypes) dans plus de dimensions qu’auparavant, nous approchant un peu plus du fonctionnement réel des systèmes biologiques dans toute leur complexité. Cependant, intégrer ces données multi-omiques massives et de nature hétérogène (qualitatives, quantitatives, temporelles ou spatiales) constitue en soi un défi pour la bioinformatique.
Ajoutons à cela les initiatives internationales telles que « The European Reference Genome Atlas » ou « Darwin Tree of Life » qui visent à produire des génomes de référence pour des dizaines voire centaines de milliers d’espèce et nous comprenons bien que le rythme auquel ces données vont s’accumuler va encore s’accélérer.
Enfin, la métagénomique, qui est l’application des techniques de séquençage à des échantillons environnementaux entiers (sol, aquatique, organe, …), révèle la diversité des microorganismes, de leurs interactions et fonctions au-delà des espèces prises individuellement.
Ainsi, ces données massives, complexes, hétérogènes et diversifiées, nous imposent un changement d’échelle et de revoir notre façon de les analyser, de les intégrer et de les comparer. En cela, l’utilisation de méthodes d’intelligence artificielle associée à l’augmentation des puissances de calcul et de stockage devrait nous permettre de relever ces défis.
Nous vivons donc en ce moment une époque absolument formidable et passionnante dans le domaine de la génomique, dopée par les progrès technologiques et les développements méthodologiques. Les hypothèses que nous pouvons tester aujourd’hui étaient simplement inimaginables il y a juste quelques années et nous promettent de belles découvertes fondamentales ouvrant de nouvelles perspectives appliquées… et encore beaucoup d’inspiration pour le travail des prochaines générations de chercheuses et de chercheurs.
