Biodiversité Temps de lecture 4 min
Création du Consortium international pangénome bovin pour explorer la diversité et le fonctionnement des génomes
Publié le 27 juillet 2023
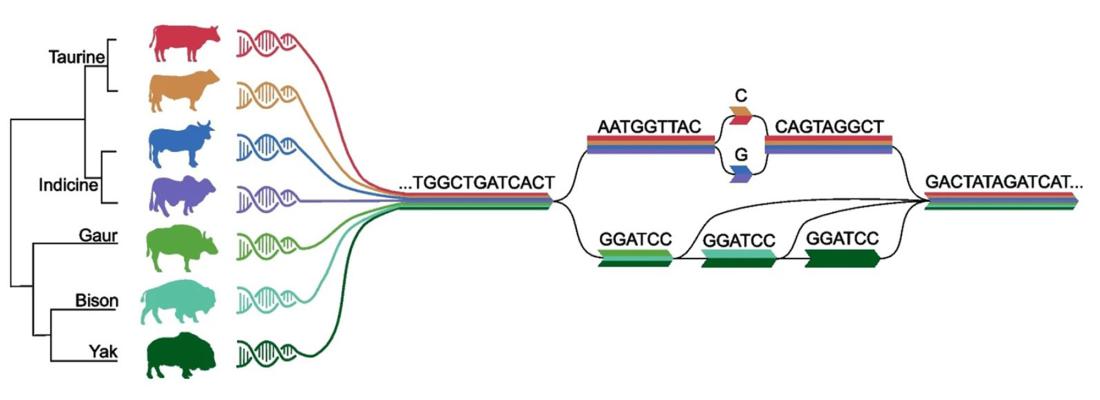
Figure. Relations entre les espèces bovines et conceptualisation du pangénome. La variation d'un nucléotide unique et la variation structurelle entre espèces/sous-espèces/races sont représentées comme des chemins différents à travers un pangénome multiespèce.
Depuis les années 2000, les travaux de génomique animale dans la plupart des espèces s’appuient sur un assemblage « de référence » du génome, c’est-à-dire la meilleure représentation possible du génome d’un individu particulier de l’espèce étudiée. Cette référence fait l’objet d’investissements importants pour en maximiser la qualité et pour l’annoter, c’est-à-dire décoder et donner un sens biologique aux informations contenues dans les séquences d’ADN. Le génome de référence est une ressource entièrement publique, facilement disponible dans les bases de données de séquences telles que Ensembl ou NCBI. La comparaison de données de séquence issues d’autres individus avec cette référence permet d’identifier les variants génétiques Cette approche très efficace a permis de caractériser au sein de chaque espèce des dizaines de millions de variants, principalement de petite taille (substitutions d’une seule base sur la séquence (SNP) et petites insertions-délétions de quelques bases à certains endroits du génome), avec les applications que l’on connait : développement d’outils de génotypage ; détection de zones du génome (QTL) ou de variants particuliers impliqués dans la variabilité de caractères d’intérêt ; sélection génomique.
Cette approche présente cependant des limitations. La recherche de variants structuraux, qui sont associés à des modifications plus importantes de la séquence génomique, est complexe à conduire et d’une efficacité limitée. Par ailleurs, de nombreuses séquences produites ne sont pas cartographiées sur la référence et sont rejetées. Cela s’explique par le fait que le génome de l’individu de référence ne contient pas forcément toutes les séquences présentes dans l’espèce et, plus généralement, par le fait que le génome de l’individu étudié diffère suffisamment de la référence pour qu’on ne puisse pas simplement caractériser leurs différences. En effet, du fait des multiples réarrangements du génome qui surviennent au cours de l’évolution d’une espèce (échange de fragments de séquence entre chromosomes, duplication ou inversion du sens de certaines portions du génome), la colinéarité des génomes n’est pas complète intra espèce, et elle l’est encore moins entre espèces, même proches.
Pour résoudre cette difficulté, il convient de considérer plusieurs assemblages, de façon à représenter toutes les combinaisons complexes de génomes présentes dans une espèce. On appelle « pangénome » la réunion de tous les génomes, c’est-à-dire à la fois les séquences communes à tous les individus (le core-génome) mais aussi toutes les séquences spécifiques à chacun. Ces assemblages ne sont pas considérés indépendamment les uns des autres, mais organisés sous la forme d’un graphe qui se prête bien à une modélisation mathématique et informatique. Dans la figure représentant un graphe d’une fraction de 7 génomes de 5 espèces proches, chacun des génomes est décrit par un chemin d’une couleur particulière.
Les chercheurs INRAE participent au Bovine Pangenome Consortium, une collaboration internationale de grande envergure qui vise à construire un pangénome rassemblant les génomes d’un grand nombre de races bovines taurines et zébus, et des espèces proches.
Un article publié dans Genome Biology précise les objectifs du consortium. Le pangénome produit sera entièrement public et accessible grâce à des outils standardisés, également proposés par le consortium. Il aura vocation à remplacer à moyen terme les assemblages de référence actuels spécifiques de race. Le consortium est ouvert et invite les partenaires qui partagent sa vision à participer à la production de ces assemblages et d'une représentation du pangénome reconnue par la communauté comme ressource publique. En permettant d’aller plus loin dans la reconstitution de l’histoire des populations bovines et dans la connaissance des liens entre l’information génomique et l’expression des caractères, cette ressource favorisera le développement d’un élevage bovin plus durable.
Référence
Smith T.P.L., Bickhart D.M., Boichard D. et al. (2023). The Bovine Pangenome Consortium: democratizing production and accessibility of genome assemblies for global cattle breeds and other bovine species. Genome Biology, 24, 139. https://doi.org/10.1186/s13059-023-02975-0
