Comprendre le développement de l’embryon des mammifères d’élevage : un atout pour la sélection des animaux de demain
Publié le 13 septembre 2024
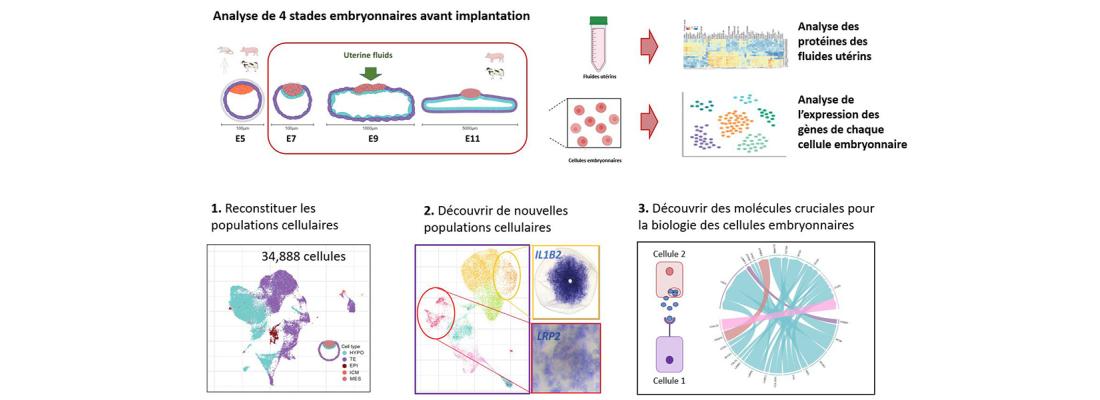
Résumé graphique de l’article scientifique. Quatre stades embryonnaires ont été étudiés, le plus précoce étant commun à l‘ensemble des mammifères et les trois suivants spécifiques aux ongulés (vache et cochon). Les fluides utérins et 35 000 cellules embryonnaires ont été caractérisés par des approches à haut débit. Ces informations ont permis de reconstituer les populations connus (1) et y découvrir de nouvelles sous-populations (en jaune et rouge), importantes pour l’implantation de l’embryon (2). Ces données ont aussi permis de mettre en évidence le dialogue moléculaire existant entre la mère et les différentes populations cellulaires de l’embryon, pour préparer l’implantation et la formation du fœtus.
Pour répondre aux enjeux des transitions agroécologique et climatique, il faut pouvoir sélectionner les animaux sur de nombreux critères souvent très hétérogènes, comme la thermorégulation, l’efficacité alimentaire, la résistance aux parasites et aux pathogènes, les problèmes de reproduction. Il faut donc que les mesures phénotypiques soient très variées. Or, certaines, comme les mesures de réponse à des infections virales, sont difficilement réalisables sur des animaux en élevage et posent des problèmes éthiques, logistiques et économiques. L’utilisation de cellules souches embryonnaires pluripotentes, dites «ES », est une piste prometteuse pour contourner ces difficultés. Les cellules ES permettent en effet d’obtenir in vitro une grande variété de lignages cellulaires représentatifs de la variété des tissus animaux, sur lesquels on peut faire de nombreuses mesures à haut-débit au laboratoire. Cependant, la culture des cellules ES chez les animaux d’élevage reste difficile à mettre en œuvre. Afin d’optimiser la dérivation de cellules ES à partir de cellules d’embryons de porc, les chercheurs INRAE, en collaboration avec une équipe de recherche allemande, ont caractérisé les cellules ES de l’embryon et les molécules nécessaires à leur survie et leur prolifération.
Pour cela, les chercheurs ont utilisé une technologie de pointe qui permet d’étudier l’ensemble des informations cellulaires issues des gènes (expression des gènes) à l’échelle de cellules uniques. Ils ont caractérisé ces informations pour 35 000 cellules embryonnaires, issues d’embryons de porcs âgés entre 5 et 11 jours après fécondation (avant l’implantation). Ils ont alors pu, par des approches statistiques et d’intelligence artificielle, identifier de nouvelles sous-populations de cellules embryonnaires, importantes pour la sécrétion de molécules nécessaires à l’implantation, qui contribuent ensuite au développement du placenta embryonnaire. Ils ont de plus observé, de façon originale, l’existence de deux types de cellules ES embryonnaires : l’une très précoce et très labile, que l’on retrouve aussi chez l’homme avant l’implantation, et une autre, plus tardive et plus stable dans le temps, qui persiste plusieurs jours avant l’implantation. La dynamique de ces deux populations cellulaires a été associée à des modifications importantes de l’activité sécrétrice de l’utérus maternel. Ainsi, grâce à l’analyse de ces 35 000 cellules, les chercheurs ont pu identifier de nouvelles molécules biologiques cruciales pour la biologie des cellules pluripotentes embryonnaires.
Ces résultats originaux permettent 1) de mieux comprendre la biologie de l’embryon précoce de mammifère, en miroir des connaissances actuelles sur les cellules embryonnaires humaines et 2) d’optimiser les conditions de dérivation et de culture des cellules pluripotentes embryonnaires porcines. Ces avancées permettront d’obtenir des lignées de cellules souches embryonnaires porcines, qui permettront, à partir d’un faible nombre d’embryons, de mieux phénotyper in vitro les populations animales et de faire le lien avec les génotypes, pour mieux sélectionner les futurs animaux de demain et ainsi répondre aux grands enjeux des transitions agroécologique et climatique.
Pour caractériser l’expression des gènes dans chaque cellule de l’embryon, les chercheurs ont utilisé la technologie dite de microfluidique en goutte. Chaque cellule est isolée dans une goutte avec des réactifs permettant de mesurer l’expression de ses gènes sans interférer avec les autres cellules. Chaque cellule est ensuite analysée pour retrouver celles qui sont semblables et les regrouper en populations cellulaires. Il est ensuite possible de comparer les populations cellulaires entre elles et d’identifier des molécules produites par leurs gènes qui sont importantes pour leur biologie.
Réf. : Adrien Dufour, Cyril Kurylo, Jan B Stöckl, Denis Laloë, Yoann Bailly, et al.. Cell specification and functional interactions in the pig blastocyst inferred from single-cell transcriptomics and uterine fluids proteomics. Genomics, 2024, 116 (2), pp.110780. ⟨10.1016/j.ygeno.2023.110780⟩
