Agroécologie 7 min
Une manip en images : CRISPR-Cas9, aussi précis que des ciseaux
Afin de montrer la réalité concrète du travail de biologie moléculaire en laboratoire, nous avons reconstitué en images une expérience de modification ciblée de gènes. Cette reconstitution permet aussi de comprendre que cette technique est très différente de la transgénèse, dans son principe et ses résultats.
Publié le 11 septembre 2015

L’objectif de la "manip"
L'objectif de l'expérience est d'explorer le potentiel des nouvelles méthodes de modifications ciblées de gènes, avec pour modèle d'étude la tomate. On cherche à lui conférer une résistance à un virus contre lequel il n'existe pas de traitement. C'est théoriquement possible en modifiant six paires de base dans un gène connu (1), ce qui revient à remplacer un allèle par un autre. Cette expérience s’inscrit dans un projet plus vaste, le projet Genius (2), qui vise à explorer la faisabilité scientifique de ces nouvelles méthodes pour améliorer des caractères d’intérêt agronomique.
Un travail de haute technicité…
Comment travaille une équipe de recherche en biologie moléculaire ? Avec quel matériel ? Avec quel degré de complexité ? Comme pour toute science expérimentale, le travail se partage entre le bureau et la paillasse, alternant les phases de conception et d'expérimentation, avec un niveau de technicité qui ne cesse de s’élever. Le biologiste moléculaire doit maitriser aussi bien des programmes d’informatique complexes que des appareils sophistiqués et surtout bien connaitre la biologie de la plante avec laquelle il travaille. Tout cela demande également une grande précision et une grande dextérité lors de l'expérimentation.
De la théorie à la pratique
Une expérimentation de ce type nécessite plusieurs mois de travail. Mais ce temps est toujours plus court que celui qu’il faudrait pour remplacer un allèle par un autre via la sélection classique par rétrocroisements successifs (3)...ce qui n'est de surcroît possible que lorsque cet allèle existe dans la diversité génétique de l'espèce, ce qui n'est pas le cas ici.
Le scénario reconstitué ici retrace les étapes clés nécessaires à la compréhension de la démarche. En réalité, s’intercalent dans ce schéma d’autres manipulations et d’autres étapes de vérifications, qui consistent par exemple à amplifier des fragments d’ADN spécifiques grâce à la technique de PCR (4), à vérifier la longueur de ces fragments en les faisant migrer sur des gels d’électrophorèse et/ou en déterminant leur séquence.
(1) Il s’agit du gène qui code pour la protéine eiF4E, un facteur d’initiation de la traduction des ARN en protéine, dont certains virus ont également besoin pour leur cycle infectieux. Avec seulement deux acides aminés modifiés, ce facteur reste fonctionnel pour la plante mais ne permet plus au virus de se multiplier dans la plante.
(2) Projet Genius : “Genome ENgineering Improvement for Useful plants of a Sustainable agriculture”, subventionné par l’ANR (2012-2019), regroupant une quinzaine de partenaires publics et privés.
(3) En particulier lorsque l’allèle recherché provient d’une plante d’une espèce éloignée, il faut de multiples croisements pour transférer cet allèle dans la plante receveuse et éliminer ensuite tous les caractères indésirables de la plante donneuse, par des rétro-croisements successifs (on croise plusieurs fois la plante ayant reçu l’allèle avec la plante parente receveuse).
(4) PCR : polymerase chain reaction, méthode d'amplification in vitro de segments d’ADN situés entre deux amorces (oligonucléotides de synthèse de 20 à 25 nucléotides homologues à la séquence matrice).
Quelques clés pour comprendre la manip
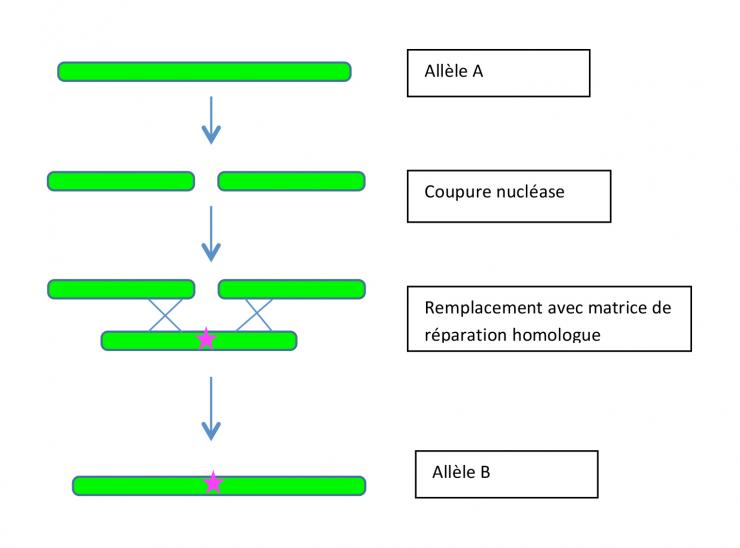
L’objectif de l’expérimentation est in fine d’obtenir une plante résistante à un virus de la famille des potyvirus, en modifiant un gène dont on connait la séquence. Il faut pour cela changer quelques paires de nucléotides dans ce gène, ce qui revient à effectuer un remplacement d'allèle (1) : allèle A remplacé par un allèle B qui confère la résistance au virus. Les techniques dites « de modification ciblée (2) » permettent aujourd’hui de faire cette modification en deux étapes : couper le gène (allèle A) à l’endroit voulu grâce à une nucléase très spécifique (3), et faire en sorte que la réparation se fasse en copiant la séquence d’ADN que l’on veut obtenir (allèle B). Pour cela, on introduit une matrice de réparation qui contient les modifications voulues. Cette matrice de réparation vient se positionner à l’endroit de la coupure grâce à son homologie de séquence.
On introduit dans la plante par biolistique (4) une « construction » qui porte donc à la fois le gène de la nucléase et la matrice de réparation. Pour avoir une quantité suffisante d'ADN correspondant à cette construction, on l’insère dans un plasmide (5) que l’on multiplie dans des bactéries.
On introduit aussi dans la construction deux gènes de sélection : un gène de résistance à un antibiotique et un gène de résistance à un herbicide pour sélectionner respectivement les bactéries, puis les plantes, qui auront intégré la construction. Seules ces bactéries et ces plantes seront capables de pousser sur un milieu contenant l’antibiotique ou l’herbicide en question.
Une fois la modification du gène réalisée, la construction est éliminée par un croisement avec une plante non modifiée. On retient parmi les descendants les plantes qui portent la modification sur le gène cible mais qui ont perdu la construction. On vérifie en serre confinée si ces plantes sont devenues résistantes au virus.
(1) Allèle : les allèles sont des formes d'un même gène qui présentent entre elles de petites variations dans leurs séquences, variations qui se traduisent par la synthèse de protéines légèrement différentes. Cette propriété est à la base de la variabilité génétique.
(2) En anglais : editing
(3) Nucléase de type Cas9-CRISPR ou TALEN.
(4) Biolistique : technique de transfert direct d’ADN par projection dans les cellules, à très grande vitesse, de microbilles d’or ou de tungstène enrobées d’ADN, par un canon à particules.
(5) Plasmide : chez les bactéries, petite molécule d'ADN circulaire, distincte de l'ADN chromosomique et capable de se multiplier de façon autonome.

Les biotechnologies vertes reposent sur un vaste ensemble de techniques de recherche en biologie végétale, techniques de culture in vitro ou techniques moléculaires telles que mutation, transgénèse, etc., dont ce dossier donne un panorama.
